Los orígenes de SARS-CoV-2: Parte 2
¿Por qué este virus infecta y se propaga tan bien entre nosotros?
Caso cortesía del Dr. Fabio Macori, Radiopaedia.org. Del caso rID: 74867
La Parte 1 de esta miniserie se centró en el lugar donde se originó el SARS-CoV-2. La Parte 2 aborda lo que lo hace diferente, y por qué es tan bueno infectando a la gente. Armados con este conocimiento, podremos abordar el miedo: ¿proviene de un laboratorio?
Una revisión de la Parte 1
Si aún no lo has leído todavía, le recomiendo que consulte la Parte 1. Escribimos sobre cómo los científicos investigan el origen de un virus. A partir de ahí, discutimos nuestra hipótesis principal para los orígenes del SARS-CoV-2. Este tiene dos partes:
La mayor parte del genoma viral probablemente proviene de un coronavirus estrechamente relacionado con un coronavirus de murciélagos de herradura llamado Bat CoV RaTG13.
El importante dominio de unión de receptores (RBD) de la proteína espiga está estrechamente relacionado con un coronavirus completamente diferente que se encuentra en los pangolines malayos.
También abordamos un resultado importante de este probable evento de recombinación: el nuevo RBD para SARS-CoV-2 es diez veces mejor que el SARS-CoV. Como recordatorio, el RBD, en parte, dicta qué tan bien la proteína espiga coloca SARS-CoV-2 en nuestras células uniendo una proteína humana en las superficies celulares llamada ACE2. El nuevo RBD muestra un aumento de 10 veces su afinidad de enlace para ACE2.
Así que antes de comenzar la Parte 2, ya tenemos una pieza del rompecabezas explicando por qué el SARS-CoV-2 es tan eficaz para infectar a los seres humanos, es muy bueno para aferrarse a la proteína humana (ACE2) necesaria para invadir nuestras células.
Cómo el SARS-CoV-2 entra en las células: Activación de la proteína Espiga
En el video de YouTube anterior, puedes ver la proteína espiga de SARS-CoV-2. Comienza con una vista de arriba hacia abajo de la proteína, luego gira alrededor, terminando de nuevo en la parte superior. Algo importante de esta proteína es que funciona en un trímero. Esto significa que tres copias idénticas de la proteína (gris claro, gris oscuro y azul, en el video anterior) se unen para realizar la función de la proteína. Al verlo girar, es posible que observe que algo se ve ligeramente raro sobre el monómero azul. Este tiene una pequeña pieza que se voltea hacia arriba en la parte superior de la proteína. De hecho, este es el RBD preparándose para unirse al ACE2 humano.
Antes de ir más lejos, aquí hay algunos datos de bioquímica rápida. Las proteínas no son estáticas, es decir, no están atascadas en la misma forma una vez que se forman. Diferentes elementos pueden hacer que modifiquen su estructura. En el caso de la espiga del SARS-CoV-2, se debe cortar una pieza de la proteína para activarla. Piense en ello como presionar el botón para abrir su paraguas. Pulsando el botón se mueve un pestillo y ¡pop! El paraguas toma su forma "activa" completa. El cortar la proteína de la espiga le permite abrirse a su forma infecciosa. Esta activación se denomina escote proteolítico (literalmente, "corte que rompe proteínas") y es necesario antes de que SARS-CoV-2 se pueda unir al ACE2 para la entrada. Muchos otros virus -como el VIH, la gripe y los virus del dengue- también deben cortar sus proteínas envoltura/espiga para activarse para la entrada viral.
Un dato más de bioquímica: las proteínas que cortan (escote proteolítico) otras proteínas se llaman proteasas. Para asegurarse no comenzar a cortar a ciegas, las proteasas solo reconocen sitios de escisión específicos en la secuencia de sus proteínas objetivo. Esta secuencia le dice a la proteasa qué proteína cortar, dónde cortarla, e incluso a veces cuándo hacerlo. Pero, ¿por qué es importante?
El arma secreta de una proteína espiga: el sitio de escisión polibásico
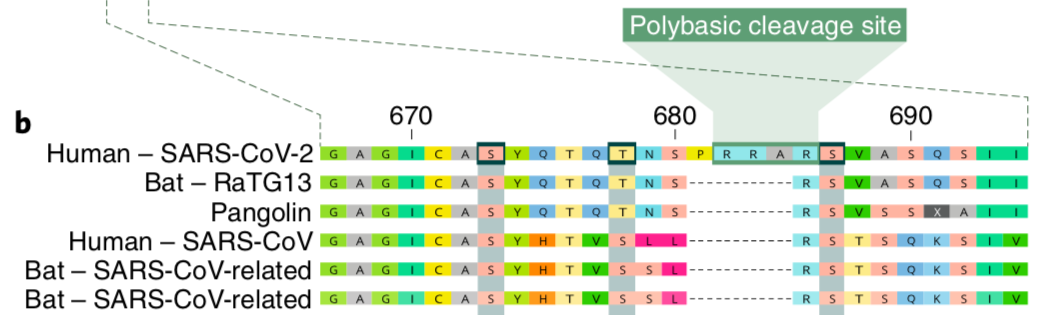
La secuencia de aminoácidos para los sitios de escisión de los virus más estrechamente relacionados con SARS-CoV-2. Editado de Andersen et al 2020.
En la imagen de arriba, se alinean las secuencias de aminoácidos (proteínas) de una pequeña región de la proteína de la espiga de seis coronavirus diferentes. Es posible que reconozca el pariente más cercano del SARS-CoV-2, el Bat CoV RaTG13, así como la secuencia del coronavirus del pangolín. Esta imagen también incluye el SARS-CoV original y dos coronavirus de murciélagos adicionales. Todo esto se hace para ilustrar un punto simple SARS-CoV-2 es la única secuencia con un sitio de escisión polibásico. Podemos ver que SARS-CoV-2 es único sin siquiera saber qué es un sitio de escisión polibásico. Donde cada uno de los otros virus tiene un vacío (líneas discontinuas), la espiga del SARS-CoV-2 tiene un trozo extra de aminoácidos (etiquetados P R R A). Estos cuatro aminoácidos adicionales resultan ser bastante importantes para el éxito del SARS-CoV-2.
El sitio de escote polibásico
El sitio de escote polibásico es un bocado. Ya discutimos que un sitio de escote les dice a las proteasas dónde cortar nuestras proteínas (y en el caso de la Espiga, esto significa que está activado). El SARS-CoV original, y otros coronavirus, tienen un sitio de escote monobásico que carece de esos aminoácidos "R" extra en el P R R A. En pocas palabras, un sitio de escisión polibásico es un objetivo más fácil de encontrar para las proteasas. Las proteínas con un sitio de escote polibásico pueden ser cortadas por la proteasa furin que se encuentra en muchos tipos de tejido. Por el otro lado, es un sitio de escote monobásico. Las proteasas que apuntan a este sitio pueden estar limitadas a un subconjunto mucho más pequeño de tejidos y ubicaciones en el cuerpo. En conjunto, esto significa:
Si la proteína espiga contiene un sitio de escisión polibásico, entonces el virus puede ser capaz de crecer y causar estragos en más tipos de tejido.
Por ejemplo, en la gripe aviar (entre muchos otros virus) donde las cepas altamente patógenas tienen un sitio de escisión polibásico, los virus pueden crecer en muchos tejidos aparte de los pulmones y el intestino de las aves, causando un aumento en la gravedad de la enfermedad (virulencia).
Aquí vemos "R" que indica el sitio de escote monobásico y "R X R/K R" que indica el sitio de escote polibásico. Las estrellas azules indican qué tejidos pueden estar infectados (modificado de Han et al. 2020).
Entonces ¿qué llevar? El sitio de escisión polibásica (es decir, activación de furin) en la proteína espiga puede aumentar la gravedad de transmisión y/o la enfermedad del SARS-CoV-2.
¿Puede el SARS-CoV-2 esconderse?
Todavía estamos aprendiendo acerca de cómo el sistema inmune humano maneja la infección por SARS-CoV-2. Una forma en que nuestro sistema inmunitario puede hacer esto es encontrar un objetivo para los anticuerpos en SARS-CoV-2. Pero, ¿qué pasaría si el virus pudiera encontrar una manera de evadir la detección? En este momento, hay algunas pruebas de que los seres humanos infectados pueden producir anticuerpos neutralizantes del virus y una sugerencia de que tal respuesta de anticuerpos podría prevenir la reinfección (al menos, en macacos). Sin embargo, no sabemos qué tan buenas son estas respuestas de anticuerpos en general. Tomemos un momento para refrescarnos sobre cómo los anticuerpos pueden funcionar para bloquear la infección.
Anticuerpos que bloquean la unión de la Espiga a ACE2, evitando la entrada de células. Creado por Christian Stevens, cortesía de BioRender.
Cuando un anticuerpo une su objetivo proteico, es increíblemente eficaz para bloquear ese objetivo. Pero, los anticuerpos no pueden luchar contra lo que no pueden unir. Los virus exitosos tienen diferentes estrategias para lidiar con esto – incluyendo "ocultar" sus proteínas objetivo de los anticuerpos (VIH) o incluso cambiar cómo se ven los objetivos con el tiempo (influenza). SARS-CoV-2 no parece estar cambiando muy rápido, así que ¿qué podría ayudar a "ocultarlo" de los anticuerpos?
Glicano ligado-O
Un glicano ligado-O es una molécula de azúcar que se une a la superficie de una proteína. En nuestros cuerpos, estos a veces se utilizan como una bandera en la proteína para indicar algo como el tipo de sangre. Otras veces ayudan a una proteína a mantener su estructura, como en los anticuerpos. Curiosamente, justo al lado del sitio de escisión polibásico en la Espiga SARS-CoV-2, también hay un nuevo sitio de glicano ligado-O.
Una hipótesis es que los glicanos crean una especie de escudo que "oculta" la proteína espiga de ser reconocida por los anticuerpos. Cuando nuestro sistema inmune busca sitios importantes para que los anticuerpos actúen, los virus que evitan esos anticuerpos tienen una ventaja selectiva. Los virus no lo hacen conscientemente (¿recuerda nuestro explicador de selección natural en la Parte 1?). A medida que los anticuerpos comienzan a bloquear todos los virus donde puedan ver el objetivo, cualquier virus que produce una mutación que resulta en un sitio de glicano ligado-O cerca del área objetivo, este lo protege de la detección por parte de nuestro sistema inmunológico. Esto se conoce como un escudo glicano.
Modificado de Karsten y Alter 2017. Los racimos amarillos indican glicanos que ocultan la columna vertebral de la proteína verde de los anticuerpos.
Entonces, ¿qué hace que el SARS-CoV-2 sea tan bueno para infectar y propagarse entre los seres humanos?
Su proteína espiga es muy buena en la unión de ACE2 humano para entrar en nuestras células (ver Parte I)
La proteína espiga se puede activar en una amplia gama de células y tejidos (sitio de escisión polibásica)
La proteína espiga podría ser capaz de ocultarse de los anticuerpos (escudo glicano)
¿Cómo y cuándo el SARS-CoV-2 desarrolló estas herramientas?
La mayor parte del genoma SARS-CoV-2 es similar a otros virus estrechamente relacionados que se encuentran en murciélagos o pangolines, por lo que entendemos los orígenes de cómo la espiga se une a ACE2 tan bien. Sin embargo, el sitio de escisión polibásico y el sitio de adición de glicano ligado-O son herramientas que solo tiene SARS-CoV-2. ¿En qué etapa podría el virus haber adquirido estas características?
Hipótesis 1: Antes de la propagación en los seres humanos
Hipótesis 2: Al amplificarse (es decir, replicarse) en humanos
Spoiler: Aún no hemos encontrado las señales para responder a esta pregunta.
Si la hipótesis 1 es correcta, entonces en algún lugar en la naturaleza, hay un reservorio de virus que se ven muy similares a SARS-CoV-2. Ya sea en pangolines, murciélagos o algún otro animal intermedio, puede haber otros virus similares al SARS que ya tienen las herramientas para propagarse en los seres humanos de nuevo. Si supiéramos que esto es cierto, podríamos tomar medidas ahora, para desarrollar programas de vigilancia y crear medicamentos y vacunas potenciales, en preparación para un evento de este tipo.
Si la hipótesis 2 es correcta, entonces cuando el SARS-CoV-2 interactuó por primera vez con los seres humanos, probablemente se reprodujo y se extendió menos bien (como hace MERS). Pero durante algunos ciclos de replicación donde el virus cometió errores, algunos mutantes ganaron el sitio de escisión polibásica y el blindaje glicano. Al hacerlo, adquirieron la ventaja en el juego de selección natural y se fueron para propagarse entre los seres humanos. Si este fuera el caso, entonces podemos implementar mejores medidas para monitorear a los humanos en busca de nuevas infecciones, y atrapar tales virus de contagio antes de que "se hagan buenos" en la propagación entre los seres humanos.
Todavía estamos en los primeros días de la investigación científica básica de SARS-CoV-2 y tomará la secuenciación de otros coronavirus en la naturaleza, antes de descubrir la historia de origen precisa de estas mutaciones particulares. Pero, descubrir la respuesta a esta pregunta ayudará a guiar nuestros planes para prevenir o manejar el próximo brote de Coronavirus antes de que alcance la gravedad que vemos con el SARS-CoV-2, hoy en día.
Para la mayoría de los científicos, la historia se detiene aquí hasta que hayamos hecho más investigación. Tenemos algunas pistas sobre los reservorios de animales, tenemos información sobre los mecanismos de transmisión y la patogénesis en los seres humanos, y tenemos algunas ideas sobre cómo responder a las próximas preguntas científicas. La mayoría de los científicos examinarán esto y dirán que estamos en el camino correcto porque:
Todos los datos recopilados hasta ahora se ajustan a nuestras hipótesis.
Nuestras hipótesis son las más parsimoniosas y satisfacen mejor la Navaja de Occam.
Nuestras hipótesis son comprobables y falsificables.
Esto implica firmemente, que el virus no fue diseñado o modificado en un laboratorio.
Sin embargo, el Internet está lleno de gente que no son científicos, y cuyas preguntas no encajan del todo en el molde científico. Y es importante considerar teorías alternativas y no simplemente descartar. En la Parte 3, examinaremos directamente algunas de las otras teorías y temores que rodean los orígenes de este nuevo coronavirus; hasta entonces, esperamos que las Partes 1 y 2 ya le hayan ayudado a armarle con respuestas científicamente rigurosas.
Estén atentos a la Parte 3, donde comenzaremos a disputar teorías de conspiración directamente ahora que tenemos las herramientas para enfrentarlas.
Amanda Kauffman
Americana-venezolana, bilingüe. Estudiante de Relaciones Internacionales. En ocasiones anteriores, ha traducido documentos para varias empresas y organizaciones en Venezuela.
akauffman00@gmail.com
Christian Stevens
Christian es un estudiante de MD/PhD en la Escuela de Medicina Mount Sinai que obtuvo su licenciatura de Harvey Mudd College.
Se unió al Benhur Lee Lab en 2018 y desde entonces ha trabajado en dos proyectos principales. El primero utiliza la ingeniería viral para explorar el uso del virus Sendai como vector viral para entregar herramientas de edición de genes. El segundo ha implicado la construcción de tuberías de construcción de trabajo computacional para el análisis utilizando la secuenciación de Illumina y la secuenciación de ARN directo de Oxford Nanopore. Los principales intereses de Christian han sido dirigir la investigación clínica de clase mundial hacia los pacientes más marginados, especialmente en los campos de las enfermedades infecciosas y la virología.
christian.stevens@icahn.mssm.edu
Twitter: @csstevens91